百家論壇更多分類
產(chǎn)品課堂
故書百回讀深思子自知——MIQE指南對qPCR實驗中核酸紫外測定的要求-下
(續(xù):MIQE指南對qPCR實驗中核酸紫外測定的要求-上)
三、實時熒光定量PCR實驗MIQE核酸質(zhì)控規(guī)則的實際運用
資料顯示,如同MIQE指南本身在國際學(xué)術(shù)期刊的文稿發(fā)表審稿環(huán)節(jié)實際貫徹落實情況的遭遇,不同期刊對文章qPCR實驗checklist的要求并不一致。整體上,按文章中對核酸質(zhì)量控制過程細節(jié)披露的詳略不同,常見3種情形。
3.1用心良苦型
皮膚淀粉樣變性(Amyloidosis cutis dyschromia, ACD)是主要發(fā)生于東亞和東南亞族群的不完全外顯的常染色體顯性遺傳疾病。糖蛋白非轉(zhuǎn)移性基因 B (glycoprotein non-metastatic gene B, GPNM-B)編碼的GPNM-B跨膜蛋白,在表皮的黑色素細胞中表達最盛。正常情況下,GPNM B蛋白與自噬蛋白和吞噬小體共定位,參與自噬蛋白被招募到吞噬小體,與溶酶體與吞噬小體融合,促進黑色素小體的形成。ACD個體發(fā)生的基因突變造成GPNMB蛋白分子截斷、在胞質(zhì)中顯著降解和錯誤細胞定位,導(dǎo)致對黑色素小體的形成、吞噬作用等負調(diào)控功能的缺失,造成 ACD中黑色素失調(diào)和淀粉樣變。對轉(zhuǎn)染野生型、突變型GPNM B基因cDNA質(zhì)粒的HeLa細胞中GPNMB mRNA表達水平檢測證實,突變體GPNMB mRNA的表達水平明顯低于野生型轉(zhuǎn)錄本。實驗中,NanoDrop 2000(相當(dāng)于NanoDrop One)被運用于以下操作:
1)從HeLa細胞提取總RNA經(jīng)DNase處理后的總RNA的純度(purity) (A260/A280、A260/A230比值)和濃度 (concentration)測定(A260)。其結(jié)果為:A260/A280≥1.9,A260/A230≥2,總RNA濃度386–466 ng/μL;
2)取1μg總RNA(約2-3μL提取液)反轉(zhuǎn)錄合成cDNA;
3) qPCR反應(yīng)的cDNA用量:上一步反應(yīng)合成的cDNA(反轉(zhuǎn)錄產(chǎn)物cDNA產(chǎn)量和濃度測定值不詳)經(jīng)10倍稀釋后,每孔取2μL稀釋cDNA模板,500 nM正、反向引物組, 2× SYBR Green PCR Master Mix在384孔PCR板上構(gòu)建10 μL反應(yīng)體系完成RT-qPCR和溶解曲線分析。
GPNM-B基因外顯子測序樣品的準備過程,同樣采用A260/A280、A260/A230比值檢驗核酸純度(A260/A280≥1.8, A260/A230≥2)、Qubit 2.0熒光染料法測定DNA濃度,還增加1%瓊脂糖凝膠電泳分析步驟以驗證DNA是否存在降解。
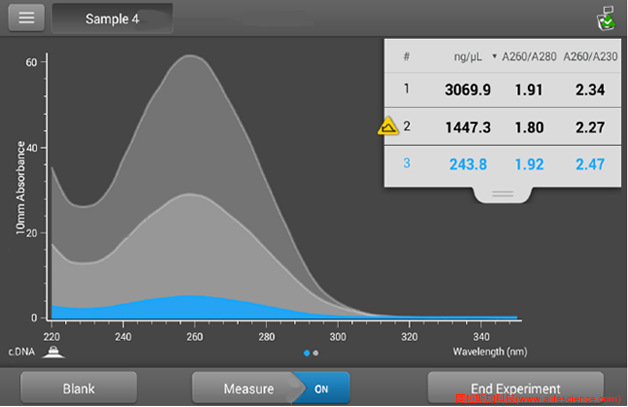
3.2中規(guī)中矩型
這部分刊文的qPCR實驗,核酸樣品質(zhì)量評估控制環(huán)節(jié),一般通過測定260/280、260/230兩項指標來評估所提取的核酸樣品的純度和濃度,測定指標的披露往往被省略。
由于細胞在3D培養(yǎng)基質(zhì)支持下懸浮生長比常規(guī)2D體外培養(yǎng)更接近在體內(nèi)真實環(huán)境生長細胞所具有的形態(tài)、增殖、分化、遷移和基因表達等特征。因此3D細胞培養(yǎng)體系統(tǒng)可用于疾病模型構(gòu)建、藥物篩選、細胞治療和再生醫(yī)學(xué)研究等領(lǐng)域。采用液滴微流控技術(shù)在單個細胞的周圍構(gòu)建起三維水凝膠基質(zhì)環(huán)境,可精確控制細胞周圍三維凝膠的沉積量。當(dāng)間充質(zhì)基質(zhì)細胞粘附于較薄凝膠時,其體積擴張得更快,具有更高膜張力和增加成骨分化功能。間充質(zhì)干細胞成骨分化標志性基因表達分析實驗中,RNA質(zhì)量評估是這的:純化的總RNA被重懸于15μL RNase-free water (注:模擬中性pH環(huán)境),NanoDrop 測定RNA 的濃度和質(zhì)量(concentration and quality);反轉(zhuǎn)錄采用Superscript-III reverse transcriptase試劑盒;每個qPCR反應(yīng)孔為50 ng cDNA用量。
文中沒有披露RNA濃度、質(zhì)量具體測定指標、測定值、反轉(zhuǎn)錄RNA用量。但給出cDNA用量,說明作者是測定cDNA的濃度后再根據(jù)吸取體積計算出cDNA含量。
另一個近紅外光免疫療法(Near-infrared photoimmunotherapy, NIR-PIT)抗腫瘤療效及機制的研究實驗中,用Nanodrop測定260/280、260/230兩項比值對腫瘤組織的總RNA純度和含量(purity and quantity)進行測評;取2μg 的總RNA用含RNase抑制劑的反轉(zhuǎn)錄試劑盒合成cDNA。
這說明實驗人員在測定總RNA的A260后,根據(jù)儀器給出濃度(μg/μL)值和移液體積計算出反轉(zhuǎn)錄總RNA用量。但文中沒有提供實時熒光定量反應(yīng)的cDNA用量,故無法得知是否進行過cDNA濃度的測定。
3.3 一筆帶過型
大量qPCR實驗論文中對核酸質(zhì)量控制過程描述都過于簡略,與MIQE指南的要求相距甚遠。
胸腺基質(zhì)淋巴生成素(thymic stromal lymphopoietin, TSLP)可通過激活2型免疫細胞、增加免Treg細胞數(shù)量,誘導(dǎo)皮膚分泌脂肪而減少白脂肪堆積,防止肥胖。2021年發(fā)表于Science上的這篇高作披露:在對皮膚18個與人類皮膚皮脂生成相關(guān)基因和TSLP基因表達檢測分析實驗中,用Nanodrop 1000測定了總RNA的含量。
另一項對新發(fā)現(xiàn)的、與外周血T細胞淋巴瘤(Peripheral T-cell lymphoma, PTCL)復(fù)發(fā)有關(guān)的嵌合轉(zhuǎn)錄本(基因融合突變而產(chǎn)生的融合基因表達的mRNA)FYN-TRAF3IP2、KHDRBS1-LCK和SIN3A-FOXO1的研究項目中,qPCR反應(yīng)前用NanoDrop 2000測定了嵌和轉(zhuǎn)錄本的濃度和純度。
磷酸甘油酸激酶1(phosphoglycerate kinase 1, PGK1)是糖酵解過程的關(guān)鍵酶。敲除pgk1基因,抑制了細胞的糖酵解反應(yīng),導(dǎo)致小分子代謝物甲基乙二醛(MGx)的積聚,進而使PGK1近端半胱氨酸和精氨酸殘基發(fā)生翻譯后的修飾,產(chǎn)生了KEAP1二聚體結(jié)構(gòu),并伴隨NRF2的積累和NRF2轉(zhuǎn)錄的激活。揭示了KEAP1-NRF2信號轉(zhuǎn)導(dǎo)通路在應(yīng)激反應(yīng)條件下的細胞代謝功能調(diào)控機制。美國斯克利普斯研究所的大V們用慢病毒載體搭載PGK1、GLO1基因靶向shRNA轉(zhuǎn)染HEK293T細胞進行的基因敲除,靶基因表達驗證實驗部分,用NanoDrop測定總RNA的濃度后取500ng-5μg用于cDNA的合成。
類似案例不勝枚舉。
可見,對實時熒光定量PCR反應(yīng)核酸質(zhì)量評估環(huán)節(jié),紫外吸光度評價法的測定指標,業(yè)界認識并不一致。評估方法上重量(Nucleic acid quantification)輕質(zhì)(Contamination assessment、Purity (A260/A280))的現(xiàn)象依然十分普遍。
四、討論
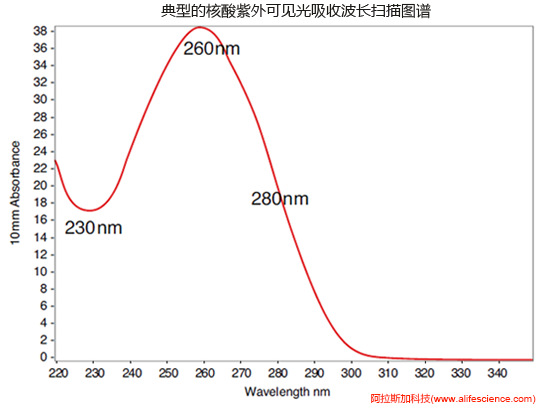
嘌呤、嘧啶、核苷、寡核苷酸或核酸(DNA和RNA)分子因共同的嘌呤環(huán)和嘧啶環(huán)共軛雙鍵而具有統(tǒng)一的260nm吸收峰特征。260nm的吸光度與所含DNA和RNA、寡核苷酸(PCR引物、探針)的濃度成正比,這是A260用于核酸定量的基礎(chǔ)。蛋白質(zhì)肽鏈的色氨酸和酪氨酸殘基具有苯環(huán)含共軛雙鍵,280nm紫外線吸收峰為其標志,故A280被用于蛋白的紫外測定和計算蛋白濃度。
在pH中性溶液中,dsDNA的A260/A280比值為1.8;ssDNA、寡核苷酸鏈和RNA的A260/A280比值為2.0。而溶液含有蛋白質(zhì)(如各種核酸酶、蛋白酶、血紅蛋白、免疫球蛋白等)或酚類殘留時,核酸A260/A280比值將低于1.8/2.0。有觀點認為:RNA樣品的A260/A280比值1.9~2.0屬高純度,1.8~1.9視為輕度蛋白污染,但還可以正常使用;但比值<1.8則視為嚴重污染,可能干擾后續(xù)實驗。對于DNA,A260/A280比值>1.9表明有RNA污染;A260/A280<1.6,視為蛋白質(zhì)/苯酚的中度污染。因此,A260測試對應(yīng)于核酸濃度(concentration)測定,而A260/280比值針對的是核酸純度(purity),用于評估吸收峰波長260nm以上的蛋白、苯酚類污染程度。
MIQE指南強調(diào)A260、A280測定須在pH中性溶液中進行(溶液的pH值改變引起核酸蛋白分子共軛體系的延長或縮短,使紫外吸收峰偏移,測定值相應(yīng)發(fā)生改變。以RNA溶液為例,酸性環(huán)境下報告的A260/A280值可能比2.0要降低0.2-0.3,而堿性環(huán)境下的測定值則相反。這是造成不同實驗報告中A260/A280、A260/A230比值不同一個不可忽視的重要因素。報告核酸樣品測定結(jié)果時,注明測試溶液的溫度和pH值更有參考價值)。眾多文獻中的做法是將核酸溶于不含DEPC、RNase的Thermo Scientific RNase-free water中檢測。而通常,25℃室溫下新鮮超純水pH值約7.0,是最方便的核酸溶劑來源。
4.1 核酸純度評估A260/A280之外是否應(yīng)引入A260/A230?
討論這一話題,是因單一A260/A280指標評估核酸純度的準確性業(yè)界一直存有爭議。
首先,因核酸對250nm-270nm范圍的消光系數(shù)比蛋白質(zhì)高很多,少量蛋白污染(如人為加入5%蛋白質(zhì))對A260/A280比值(處于1.96-1.99區(qū)間)影響有限。只有當(dāng)?shù)鞍踪|(zhì)肽鏈中色氨酸殘基(吸收峰為280nm)比例較高,蛋白的混入才會出現(xiàn)A260/A280比值顯著改變。純蛋白樣品A260/A280比值為0.57,而只需少量核酸混入即可使比值迅速躥升至1.0上下。因此,A260/A280比值最適于對蛋白制品中核酸污染評估,作為指示核酸樣品中蛋白污染程度并不靈敏。
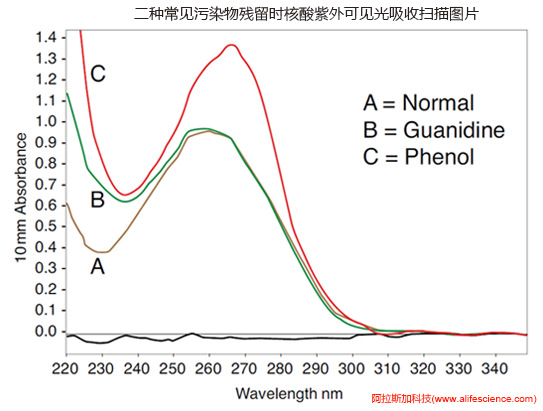
其次,苯酚、異硫氰酸胍、PEG等提取試劑、雜質(zhì),在230nm、260nm、280nm波長附近均有不同程度光吸收,增加A230、A260和A280測定值,會干擾A260/A280比值。譬如苯酚殘留,不僅使A230、A260和A280讀數(shù)上升。同時,因其吸收峰與核酸吸收峰合并后整體遷移移至270nm附近。表面上樣品 260/280 比值接近正常或降低,但樣品吸收峰出現(xiàn)在270nm,且存在260/230比值降低現(xiàn)象,實際存在苯酚殘留。再如胍鹽、糖原和碳水化合物等,有230nm的吸收峰,雖不影響A260、A280讀數(shù)及260/280比值,但會壓低260/230比值。此時樣品260/280值達標,但260/230<1.6,污染是存在的。
盡管因DNA、RNA樣品A260/A280比值不同,理論上,基因組DNA殘留會使RNA的比值低于2.0。但基于上述各種復(fù)雜情況,僅憑A260/A280比值是無法有效確認樣品提取過程中基因組DNA的污染及嚴重程度的。因此,單一A260/A280比值實現(xiàn)對核酸純度,特別是核酸抑制劑污染的評估,是不全面的。
克服A260/A280單一指標缺陷的有效辦法是核酸樣品波長掃描分析。以RNA為例,純RNA掃描得到的是一平滑曲線:有A230波谷、A260波峰,A320\A340貼近背景基線;A260/A280≈2.0,A260/A230≈2.0,A260/A215≈1.0。
部分固定波長的微量紫外光度計,如賽默飛的NanoDrop Lite Plus微量紫外光度計(A230/A260/A280),無法進行全波長掃描,可用A260/A280、A260/A230雙比值來評估污染物的殘留情況。
事實上,本文所引文獻中就有實驗采用的是A260/A280 +A260/A230雙比值法。這比SPUD抑制劑測定法,顯然更加快捷、經(jīng)濟易行。
4.2 如何評估RNA樣品中基因組DNA污染?
qPCR實驗中,反轉(zhuǎn)錄合成的cDNA與殘留的基因組DNA都可作為PCR模板被擴增,特別是采用SYBR Green染料標記的測試中,殘留的DNA熒光信號污染會影響定量數(shù)據(jù)準確性。因此,去除總RNA中的基因組DNA污染,對于采用RT-qPCR方法進行基因表達定量分析具有特殊必要性。
而完全依賴A260/A280單一指征對核酸污染評估確有潛在風(fēng)險。
由于基因組DNA分子量通常比RNA分子量大得多,在凝膠電泳中遷移率存在明顯差異,因此可通過瓊脂糖凝膠電泳予以有效分離和鑒定。通常,總RNA中如有基因組DNA殘留,DNA條帶因為遷移緩慢而滯后于所有RNA條帶,在凝膠上表現(xiàn)為在28S rRNA帶上方、接近上樣孔附件出現(xiàn)新的彌散條帶。MIQE指南規(guī)定,須記錄實驗中對RNA樣品經(jīng)何種DNase酶及處理條件這一細節(jié)。同時建議實驗者提供反轉(zhuǎn)錄操作前樣品的凝膠電泳分析證據(jù)。
增加瓊脂糖凝膠電泳分析,不僅簡便易行,還增進核酸質(zhì)量評估的嚴謹性和測試結(jié)果可靠性。盡管在MIQE指南屬于核酸提取操作中D類審查事項,但本文所引用多篇刊文的實驗中都采用了該方法。
A260/A280比值之外,MIQE指南要求記錄(測試所用具體方法流程)可耐受基因組DNA污染含量的閾值(the threshold cutoff criteria for the amounts of such contamination that are tolerable.),并報告每個測試靶標核酸樣品孔、陽性對照樣品(內(nèi)參對照基因)孔和無逆轉(zhuǎn)錄對照孔(只有RNA而無cDNA,可檢驗RNA樣品在經(jīng)DNase處理后中是否還有基因組DNA的殘留)的Cq比較結(jié)果(the results from a comparison of Cqs obtained with positive and no-reverse transcription controls for each nucleic acid target.)以證明是否存在基因組DNA污染干擾情形。
4.3 簡便易行的核酸樣品PCR抑制劑評估建議
核酸質(zhì)量評估控制環(huán)節(jié)還有一審查事項就是PCR抑制劑污染的評估。
無論是MIQE指南與人們實際工作中執(zhí)行的做法是一致的,即采用核酸樣品連續(xù)倍比稀釋,根據(jù)qPCR標準曲線所計算的PCR擴增效率來證明是否存在PCR抑制劑污染。
A260/A280 +A260/A230雙比值,可視為PCR抑制劑評估的間接證據(jù)。
4.5 MIQE指南紫外吸光度法核酸質(zhì)量評價環(huán)節(jié)是否有值得商榷之處?
首先,MIQE checklist表中反轉(zhuǎn)錄步驟,用Amount of RNA(RNA質(zhì)量,即μg或ng質(zhì)量數(shù))及Priming oligonucleotide concentration都屬于必須報告內(nèi)容,對qPCR反應(yīng)cDNA/DNA模板用量不作要求,這不合理。因為cDNA轉(zhuǎn)錄效率存在一定程度的基因特異性、反應(yīng)時間依賴性和各廠家試劑盒合成效率差異化的事實,相的總RNA用量、RT酶和RT時間,cDNA合成產(chǎn)量存在差異是完全可以預(yù)料的,這勢必導(dǎo)致稀釋后上樣的cDNA輸入量不同,再考慮到不同qPCR儀擴增性能不一致,不明確cDNA起始用量,實驗結(jié)果的可重復(fù)性和穩(wěn)定性就是空話。而現(xiàn)實情況是,大量刊文中都標明qPCR反應(yīng)起始核酸模板用量的做法。
其次是關(guān)于核酸品質(zhì)紫外評估方法的問題。不僅A260/A230比值是否應(yīng)該引入問題。對于Contamination assessment (DNA or RNA)與Purity (A260/A280)的內(nèi)涵與聯(lián)系,MIQE也欠一個說明的。從指南原文看,核酸污染評估指的是一種核酸(如RNA)中有另一種核酸(如基因組DNA)的少量殘留污染,并且認為A260/A280比值(Purity測定指標)下降可認定RNA樣品中存在DNA污染。如果Contamination assessment(E類)可用A260/A280比值變化(D類)來準確評估,那把二者分別劃為E、D,本身自相矛盾。除非諸如從微量紫外可見光度計吸光度測定、瓊脂糖凝膠電泳、熒光染料法、Agilent 2100 Bioanalyzer, Qiagen QIAxcel等中找到了比A260/A280比值更可靠、簡便、經(jīng)濟的方法,否則,作為一個標準體系,Contamination assessment (DNA or RNA)的可執(zhí)行性、嚴肅性難免要大打折扣。
目前無論是綜合應(yīng)用類常規(guī)紫外可見分光光度計(如日立UH5300等),抑或生物學(xué)核酸蛋白測定用紫外可見光度計(如eppendorf BioPhotometer Plus、NanoDrop One系列、Implen NanoPhotometer N50及Nano 100/Nano 300等),系統(tǒng)預(yù)置的核酸測定工作協(xié)議中,A230、A260、A280、A260/A230A和260/A280指標早已用作行業(yè)標準。
足見在核酸質(zhì)量評價具體指標的問題上,MIQE指南這把寶刀的確有個豁口。
參考文獻
[1]Fleige S, Pfaffl MW. RNA integrity and the effect on the real-time qRT-PCR performance. Mol Aspects Med 2006;27:126 –39.
[2]Nolan T, Hands RE, Bustin SA. Quantification of mRNA using real-time RT-PCR. Nat Protoc 2006;1:1559–82.
[3]Nolan T, Hands RE, Ogunkolade BW, et al. SPUD: a qPCR assay for the detection of inhibitors in nucleic acid preparations. Anal Biochem 2006;351:308–10.
[4]Stephen A Bustin, Vladimir Benes, Jeremy A Garson, et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem,2009;55(4):611-22.
[5]High Capacity cDNA Reverse Transcription Kit User Guide (Revision A.0)
[6]Bio-Rad iScript cDNA Synthesis Kit Product Insert (Ver D)
[7]Chi-Fan Yang, Shuan-Pei Lin, Chien-Ping Chiang, et al. Loss of GPNMB Causes Autosomal-Recessive Amyloidosis Cutis Dyschromica in Humans. Am J Hum Genet. 2018 Feb 1; 102(2): 219–232.
[8]Sing Wan Wong, Stephen Lenzini, Raymond Bargi, et al. Controlled Deposition of 3D Matrices to Direct Single Cell Functions. Adv Sci (Weinh) 2020 Oct; 7(20): 2001066.
[9]Tadanobu Nagaya, Jay Friedman, Yasuhiro Maruoka, et al. Host immunity following near-infrared photoimmunotherapy is enhanced with PD-1 checkpoint blockade to eradicate established antigenic tumors. Cancer Immunol Res. 2019 Mar; 7(3): 401–413.
[10]Ruth Choa, Junichiro Tohyama, Shogo Wada, et al. Thymic stromal lymphopoietin induces adipose loss through sebum hypersecretion. Science. 2021 Jul 30; 373(6554): eabd2893.
[11]Koen Debackere, Lukas Marcelis, Sofie Demeyer, et al. Fusion transcripts FYN-TRAF3IP2 and KHDRBS1-LCK hijack T cell receptor signaling in peripheral T-cell lymphoma, not otherwise specified. Nat Commun. 2021; 12: 3705.
[12]Michael J. Bollong, Gihoon Lee, John S. Coukos, et al. A metabolite-derived protein modification integrates glycolysis with KEAP1-NRF2 signaling. Nature. 2018 Oct; 562(7728): 600–604.
[13]MIQE指南及qPCR及RT-qPCR前的核酸評估
[14]NanoDrop在核酸的質(zhì)控中的應(yīng)用
[15]NanoDrop 在Real-time PCR 中的應(yīng)用
[16]NanoDrop One微量紫外/可見光分光光度計用戶手冊(Rev. B)















